05.08.2016: 3 Funde unterwegs gemacht
Liebe Pilz-Freunde,
auf dem Weg von A nach B bin ich wie immer mal hier, mal da stehen geblieben. Wieder fand ich viele Pilze - die 3 schönsten Funde zeig ich Euch...
Los geht's...
Fundnummer: 2016-08-05-1505
Morphologische Daten:
Fundort: ca. 550 müNN. ca. N50, O12, auf steinigen, sandigen und lehmigen Boden unter einer Weide
Fundzeit: 05.08.2016
Wuchsform: einzeln
Sporenkugel: 11 mm, graubraun
Scheitelöffnung: graubraun gefranst bis gewimpert, Durchmesser ca. 1 mm
Peristom-Hof: nicht vorhanden
Sternlappen: 9 Stück, beige, mit minimalem rosa-stich
Größe: Durchmesser ca. 2,5 cm,
Sporenpulverfarbe: nicht beurteilt
Geruch: pilzig
Geschmack: nicht probiert
Gewimperter Erdstern (Geastrum fimbriatum):


Fundnummer: 2016-08-05-1541
Morphologische Daten:
Fundort: ca. 550 müNN. ca. N50, O12, auf Rasen (Garten)
Fundzeit: 05.08.2016
Wuchsform: gesellig
Hutform: konvex bis stumpfkegelig, manchmal mit leichter Papille
Huthaut: dunkelbraun, nach außen hin heller werdend, radialfaserig, Lamellen am Rand durchscheinend, im Zentrum haarfilzig
Hygrophanität: ja, dadurch radiale helle Streifen entstehend
Hutrand: kantig, bei einem jungen Exemplat etwas eingerollt, bei alten Exemplaren schartig
Lamellen: graurosa, Zwischenlamellen
Lamellenschneiden: bauchig, sehr fein behaart (Lupe)
Lamellen-Stielübergang: sehr tief ausgebuchtet angewachsen, am Stiel leicht herablaufend
Stiel: grau-braun, oben heller, kahl, unten etwas bereift, fest und steif aber hohl
Stielbasis: knollig, mit weißem Mycelfilz
Fleisch: ohne Besonderheiten
Größe: Hutdurchmesser ca. 1,3-2 cm, Stiellänge ca. 4 cm, Stieldurchmesser ca. 1,5-2 mm
Sporenpulverfarbe: rosa
Geruch: mehlig
Geschmack: nicht bewertet
Mikroskopische Daten:
HDS-Präparat:
Schnallen: keine gefunden
leptonioide Körnchen im HDS-Präparat: vorhanden
Epikutis: flach liegende, dicht gepackte, zylindrische Hyphen mit leicht verdicktem Ende, Pigment intrazellulär (auch klumpig intrazellulär) und manchmal etwas epimembranär inkrustiert, keine Schnallen gefunden
Breiten der Epikutis-Hyphen: ca. (2,9) 4 - 6,7 (9) µm
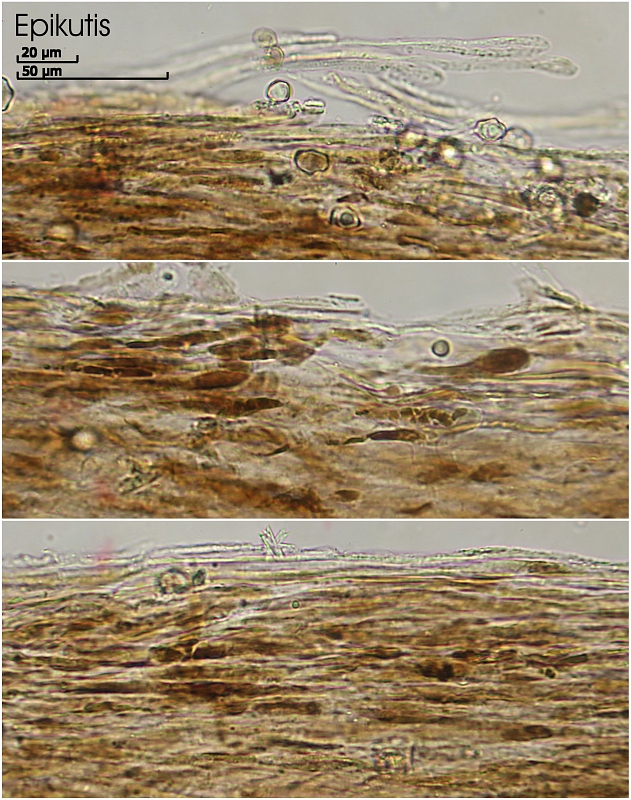
Subkutis: hyaline Hyphen, zylindrisch, in weitem Abständen inkrustiert (was man nicht als Inkrustierung im allgemeinem Sinn deuten kann), keine Schnallen gefunden
Breiten der Subkutis-Hyphen: ca. (1,8) 4 - 13 (18) µm

Lamellen-Präparat:
Schnallen: keine gefunden (mit Ausnahme an 2 Cheilozystiden)
leptonioide Körnchen im Lamellen-Präparat: vorhanden

Cheilozystiden:
keulig bis spindelförmig (am häufigsten), auch ampullenförmig, auch gliedrig septiert, grünlich inkrustiert, an 2 Cheilozystiden waren an der Basis Schnallen vorhanden
Messwerte Cheilozystiden:
Präparat: Lamellenstück (Exsikkat) --> Ammo 10%; Untersuchungsmedium: GSM; Messwertanzahl: n = 16
Test auf Normalverteilung nach Anderson Darling: Länge: normalverteilt; Breite: normalverteilt; Q: nicht normalverteilt
Standardabweichung S. D.: von L × B: 8,3 × 2,3 µm; von Q: 1,6
Median: von L × B: 44,5 × 10,4 µm; von Q: 4,2
Arithmetischer Mittelwert Me: von L × B: 45,8 × 10,7 µm; von Q: 4,5
Abmessungen nach Quantil-Verfahren mit 80%-Standardbereich: für L × B: (35,3) 36,3 - 55,2 (67,3) × (7) 8,5 - 13,8 (16) µm; für Q: (2,2) 3,5 - 5,1 (9,7)

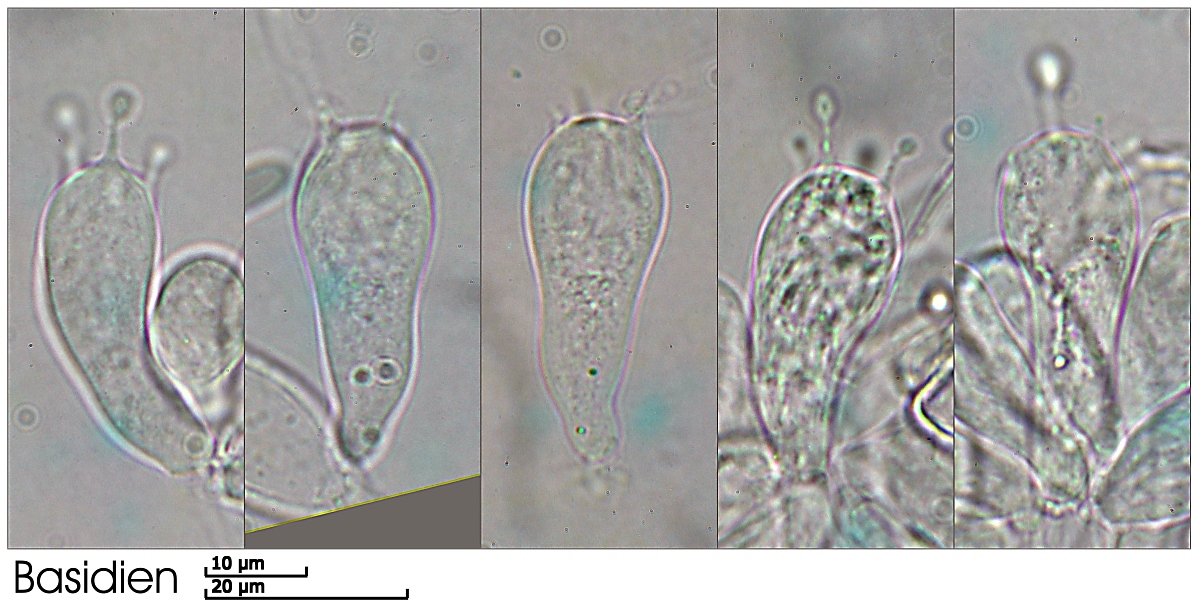
Basidien:
4-sporig, keulig, keine schnellen gesehen aber das mag nichts heißen
Maße: ca. 31 - 37,4 × 11 - 14,4 µm
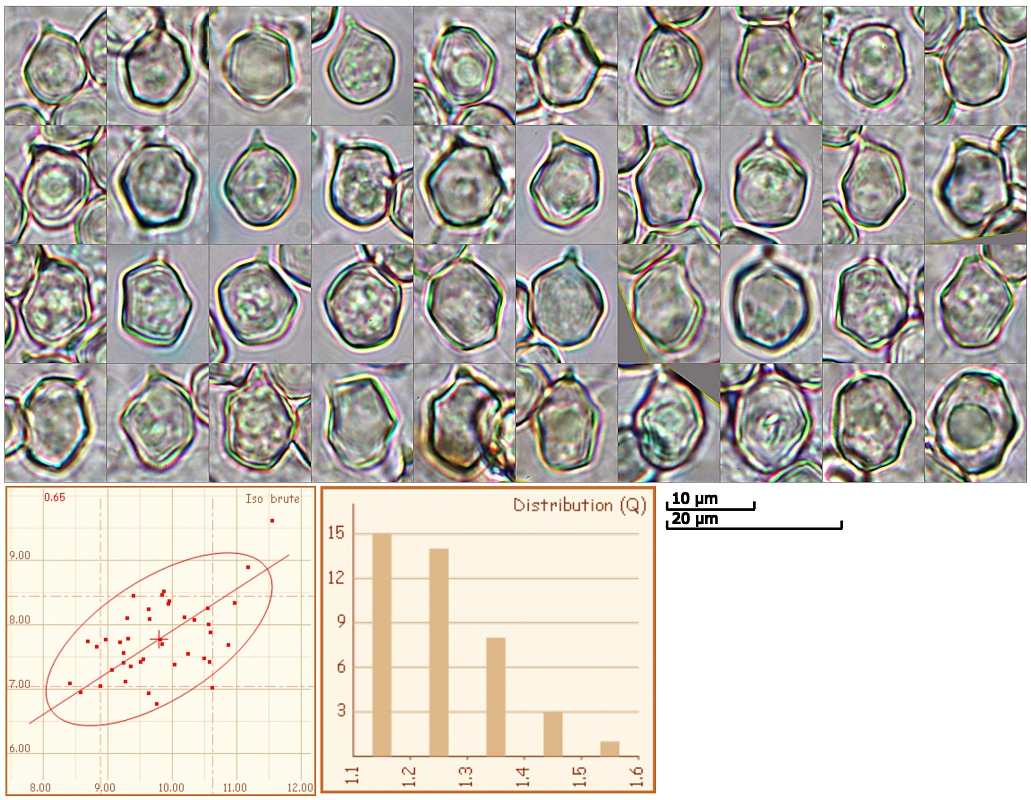
Sporen:
variabel isodiametrisch (am häufigsten – siehe q-Distribution-Diagramm) bis schwach hetereodiametrisch, 5 oder 6-eckig, Ecken nicht ausgeprägt
Maße:
Präparat: aus Lamellenstück (Exsikkat) ausgewaschen; Untersuchungsmedium: GSM; Messwertanzahl: n = 41
Test auf Normalverteilung nach Anderson Darling: Länge: normalverteilt; Breite: normalverteilt; Q: normalverteilt
Standardabweichung S. D.: von L × B: 0,7 × 0,6 µm; von Q: 0,1
Median: von L × B: 9,8 × 7,7 µm; von Q: 1,3
Arithmetischer Mittelwert Me: von L × B: 9,8 × 7,8 µm; von Q: 1,3
Abmessungen nach t-Verteilungsverfahren mit 80%-Konfidenzintervall: für L × B: (8,4) 8,8 - 10,8 (11,6) × (6,8) 7 - 8,5 (9,6) µm; für Q: (1,1) 1,1 - 1,4 (1,5)
Abmessungen nach t-Verteilungsverfahren mit 90%-Konfidenzintervall: für L × B: (8,4) 8,6 - 11 (11,6) × (6,8) 6,8 - 8,8 (9,6) µm; für Q: (1,1) 1,1 - 1,4 (1,5)
Schlüssel-Weg:
Gröger Teil I - S. 478
A > A* > B > B* > C > C* > G > H > Teilschlüssel d bis l S. 513 > e > 1b > 3 > 3a > 4 > 4b > 6 > 6b > Entoloma pratulense
Nachprüfung anhand "Entoloma (Basidiomycetes, Agaricales) of alpine habitats on the Hardangervidda near Finse, Norway, with a key including species from Northern Europe and Greenland": Geruch war bei meinem nicht neutral, Schnallen waren bei mir nur an den Cheilozystiden zu finden, ansonsten keine Widersprüche.
Aber: Nachprüfung nach FE5a: Cheilozystiden Form passt nicht, Habitus (vor allem Stiel) anders
A > A* > B > B* > C > C* > G > H > Teilschlüssel d bis l S 513 > e > 1b > 3 > 3a > 4 > 4b > 6 > 6b > Entoloma langei nachprüfen: Cheilozystiden Form passt nicht
A > A* > B > B* > C > C* > G > H > Teilschlüssel d bis l S 513 > d > 1 > 1b > 6 > 6a 7 > 7b > 8 > 8b (Geruch im Schlüssel falsch) 9 > 9b > 10 > 10a > Entoloma tenellum (Cheilos anders)
Pilzkompendium 360: Sporen mit ausgeprägten Ecken stimmt nicht, Cheilozystidenform stimmt nicht --> kann nicht sein.
A > A* > B > B* > C > C* > G > H > Teilschlüssel d bis l S 513 > e > 1a > 2 > 2b > Entoloma globuliferum --> passt nicht
A > A* > B > B* > C > C* > G > H > Teilschlüssel d bis l S 513 > e > 1b > 3 > 3a > 4 > 4a > 5 > Entoloma canosericeum
Der "deutlich befaserte Stiel" passt nicht, die Stieldicke passt nicht, "trocken stark befasert" passt nicht, "saprotroph im Laubwald" passt nicht, Habitus (vergleich mit Dähncke) passt nicht.
A > A* > B > B* > C > C* > G > H > Teilschlüssel d bis l S 513 > f > 1 > 1b (eher nicht) > 2 > 2b > 5 > 5a > 6 > Entoloma kerokarpus (schnallen fehlen)
Pilzkompendium 373 - Hutbeschreibung stimmt nicht, Lamellenfarbe passt nicht, Stielform passt nicht, Stielbereifung passt nicht --> kann nicht sein.
A > A* > B > B* > C > C* > G > H > H* > Teilschlüssel a bis c S 486 > Teilschlüssel a > 1 > 1b > 2 > 2b > 5a > 6 > 6b > 8 > 8a > Entoloma pallideradicatum --> passt nicht
A > A* > B > B* > C > C* > G > H > Teilschlüssel d bis l S 513 > l > 1b > 2 > 2b > 3 > 3b > 4 > 4b > Entoloma fernandae --> passt nicht
FN II - Seite 517:
1 > 2 > 3 > 4 > 5 > 6 > 9 > 10 > 11 > 12 > Key L p. 569 > 1 > 3 (Schnallen zwar wenig aber eben da) > 4 > Entoloma pratulense
1 > 2 > 3 > 4 > 5 > 6 > 9 > 10 > 11 > 12 > Key L p. 569 > 1 > 3 (Schnallen zwar wenig aber eben da) > 4 > Entoloma langei --> siehe oben - kann nicht sein
1 > 2 > 3 > 4 > 5 > 6 > 9 > 10 > 11 > 12 > Key L p. 569 > 1 > 2 > Entoloma tenellum --> siehe oben - kann nicht sein
1 > 2 > 3 > 4 > 5 > 6 > 9 > 10 > 11 > Key J p. 553 (eher nicht) > 1 > 4 > 9 > 10 > 11 > 12 > 13 > 14 > 17 > 18 > 30 > 31 > 32 > Sackgasse
1 > 2 > 3 > 4 > 5 > 6 > 9 > 10 > 13 (eher nicht) > 14 > Key O p. 575 > Sackgasse
Ein optisch sehr ähnliche Kollektion ist hier zu sehen:
http://www.pilzepilze.de/cgi-bin/webbbs/parchive2011.pl?noframes;read=215782
Nach Prüfung aller alternativen bleibt nur eine Möglichkeit:
der Almen-Glöckling (Entoloma cf pratulense) ??. Wegen der Unsicherheit führte ich eine Sequenzanalyse durch.
Leider schlug die PCR mit den Standard-Rückwärtsprimer fehl, sodass nur ein unvollständiges Stück der ITS aus der Vorwärtsprimer-PCR vorhanden war. Das machte aber nichts, da ich ja kein Phylogramm erstellen, sondern eine eventuelle Bestimmung erzielen wollte. Nach Abgleich der ITS-Sequenz mittels Divergenzmatrix ergab sich folgende finale Sequenz:
>2016_08_05_1541_Entoloma_spec
AGGTGAACCTGCGGAAGGATCATTATTGAATTAAACGTGGGATGGGTTGATGCTGGCTTGTTCAAAGCATGTGCACATCCATTCTATCTTTAAACCCTCCTGTGCACCTTGTGTAGGCCTTGAACGAACAATCTGTCGAAGGGAAATCACTCTTCGGTGACCGAGACTGCTGCTGCATTTGCACAGCTGTTTTGTATATGTTCTTTCAAGGTTCTATGTTTCCCATTATAAAATACTGTAAAAGAATGTCCTTTTTATTGGCCTTCTGTGCCTTTAAACATAAAATACAACTTTCAACAACGGATCTCTTGGCTCTCGCATCGATGA
Diese hat leider nur Identität mit einer einzigen Sequenz, nämlich 100% Identität mit LN850612.1 Entoloma velenovskyi.
Nun waren die Fragen:
a) kann Entoloma velenovskyi so kleine Sporen haben?
b) Ich habe keine Vergleichssequenz für Entoloma pratulense. kann die ermittelte Sequenz Entoloma pratulense sein?
Hierzu fragte ich natürlich Machiel Noordeloos, der mir mitteilte:
"Es ist ja schon dass es gelungen ist ITS zu sequenzieren. Aber: was ist es. Falls es 100 % identische ist mit E. velenovskyi in Genbank/Unite, dann bedeutet es, dass es identisch ist mit Entoloma velenovskyi sensu Kokkonen, basiert auf einer identification von A. hausknecht, aber nicht perse von den Typus des Artes. Dieser Match sagt also wesentlich nichts...." "...Deiner Art ist deswegen noch immer Namenlos, bis wir einer Typus sequenz haben, oder, was in diesem Fall wahrscheinlicher ist, wir ein sequenz haben vom einer Epitypus, da das Typus von Velenovsky ist Alkoholmaterial in sehr schlechter Zustand und wird nie noch brauchbares DNA geben."
Damit bleibt der Fund vorerst unbestimmt, bzw. momentan nur der Almen-Glöckling (Entoloma cf pratulense):




Fundnummer: 2016-08-05-1600
Morphologische Daten:
Fundort: ca. 550 müNN. ca. N50, O12, auf Rasen
Fundzeit: 05.08.2016
Wuchsform: gesellig
Hutform: halbkugelig
Huthaut: ockergelb, kahl
Hygrophanität: nicht festgestellt
Hutrand: ohne Besonderheiten
Lamellen: braun, bauchig, mit Zwischenlamellen
Lamellenschneiden: weißlich
Lamellen-Stielübergang: angeheftet
Stiel: ockergelb, wie ein Strohhalm aussehend, längsfaserig, fest und steif, manchmal verdreht, hohl, mit Mark gefüllt
Stielbasis: knollig
Fleisch: ockergelb
Größe: Hutdurchmesser ca. 2-3 cm, Stiellänge ca. 3-5 cm, Stieldurchmesser ca. 1,5-2,5 mm
Sporenpulverfarbe: braun
Geruch: erdig und mehlig
Geschmack: nicht probiert
Das ist der Halbkugelige Ackerling (Agrocybe pediades):



Das war's für heute....
Zurück zur Schwammer-Übersicht